
Abstrak
Kekhawatiran tentang penerapan pengujian farmakogenetik (PGx) di berbagai kelompok keturunan telah meningkat akibat kurangnya representasi populasi non-Eropa dalam penelitian PGx. Panel PGx saat ini mungkin gagal mendeteksi varian yang relevan pada populasi non-Eropa, sehingga meningkatkan kemungkinan hasil negatif palsu. Untuk menyelidiki hal ini, kami menilai frekuensi alel referensi (*1) dan genotipe (*1/*1) berdasarkan keturunan yang dilaporkan sendiri dalam kelompok yang terdiri dari 1.086 remaja berusia 6–24 tahun yang menjalani pengujian PGx. Pengujian mencakup 10 farmakogen ( CYP2B6 , CYP2C19 , CYP2C9 , CYP2D6 , CYP3A4 , CYP3A5 , NUDT15 , SLCO1B1 , TPMT , dan VKORC1 ) menggunakan panel yang mencakup semua alel Asosiasi Patologi Molekuler Tingkat 1 dan 53% alel Tingkat 2. Dibandingkan dengan orang Eropa ( n = 727), orang non-Eropa ( n = 359) memiliki frekuensi alel *1 yang lebih tinggi untuk CYP2C9 , CYP2D6 , dan CYP3A5 (semua P < 0,01), sementara orang Eropa memiliki frekuensi yang lebih tinggi untuk CYP2C19 dan VKORC1 (semua P < 0,01). Demikian pula, frekuensi genotipe *1/*1 lebih tinggi pada orang non-Eropa untuk CYP2C9 dan CYP3A4 (semua P < 0,01), tetapi lebih tinggi pada orang Eropa untuk VKORC1 ( P < 0,01). Estimasi negatif palsu melebihi 1% untuk CYP2B6 , CYP2D6 , CYP2C9 , CYP2C19 , dan SLCO1B1 pada setidaknya satu kelompok leluhur. Temuan ini mendukung gagasan bahwa frekuensi alel *1 dan genotipe *1/*1 lebih sering terjadi pada orang non-Eropa untuk gen tertentu, tetapi orang Eropa juga berisiko sebanding untuk hasil negatif palsu. Cakupan alel yang diperluas pada panel PGx dapat mengurangi risiko negatif palsu, meningkatkan kesetaraan dalam pengujian PGx di seluruh populasi yang beragam.
Sorotan Studi
APA PENGETAHUAN TERKINI TENTANG TOPIK TERSEBUT?
Pengujian farmakogenetik (PGx) sering kali mengecualikan varian genetik yang umum pada populasi non-Eropa, yang menyebabkan potensi hasil negatif palsu. Panel PGx saat ini bervariasi dalam cakupan alel, dan penerapan klinisnya di berbagai kelompok leluhur masih belum dieksplorasi.
PERTANYAAN APA YANG DIJAWAB OLEH STUDI INI?
Apakah frekuensi alel *1 dan genotipe *1/*1 berbeda dalam populasi Eropa dan non-Eropa, dan apakah perluasan cakupan alel berdampak pada frekuensi negatif palsu?
APA YANG DITAMBAHKAN STUDI INI PADA PENGETAHUAN KITA?
Frekuensi alel *1 dan genotipe *1/*1 bervariasi menurut kelompok keturunan dan farmakogen, dengan perkiraan frekuensi negatif palsu di atas 1% untuk beberapa farmakogen pada kelompok non-Eropa dan Eropa—menyoroti pentingnya perluasan cakupan panel PGx untuk mengurangi risiko negatif palsu di seluruh populasi yang menjalani pengujian PGx.
BAGAIMANA HAL INI DAPAT MENGUBAH FARMAKOLOGI KLINIS ATAU ILMU TRANSLASIONAL?
Temuan ini mendukung perlunya standarisasi dan perluasan cakupan panel PGx agar mencakup berbagai varian genetik. Perubahan ini dapat mengurangi hasil negatif palsu, meningkatkan kesetaraan pengujian PGx, dan meningkatkan strategi pengobatan yang dipersonalisasi di berbagai populasi.
Pengujian farmakogenetik (PGx) telah muncul sebagai alat klinis, yang memungkinkan pemilihan dan pemberian dosis obat yang dipersonalisasi berdasarkan profil genetik individu. 1 Namun, muncul kekhawatiran tentang penerapan pengujian PGx di berbagai kelompok leluhur/etnis. Kekhawatiran ini muncul dari fakta bahwa penelitian PGx terutama dilakukan pada orang-orang keturunan Eropa. Dengan demikian, panel pengujian PGx mungkin tidak menangkap varian genetik pada populasi non-Eropa, dan selanjutnya meningkatkan kemungkinan hasil uji negatif palsu pada populasi tersebut. 2 , 3
Hasil uji PGx negatif palsu, jika ada, paling sering melibatkan alel *1. Alel *1 adalah alel referensi atau alel “tipe liar”—ditetapkan saat tidak ada alel yang termasuk dalam panel PGx yang terdeteksi. Alel *1 diasumsikan memiliki fungsi normal, dan yang ditetapkan sebagai genotipe *1/*1 diasumsikan memiliki fenotipe “normal”. Dengan demikian, keyakinan pada hasil fenotipe “normal” akan sering bergantung pada cakupan alel panel PGx yang digunakan. Sayangnya, pengujian PGx tidak terstandarisasi, yang berarti cakupan alel bervariasi dari satu laboratorium ke laboratorium lainnya. 4 – 6
Untuk mengatasi variabilitas dalam pemilihan alel di berbagai laboratorium, Asosiasi Patologi Molekuler (AMP) mengembangkan pedoman pemilihan alel. Pedoman ini mengklasifikasikan alel ke dalam kategori Tingkat 1 (set minimum) dan Tingkat 2 (opsional) berdasarkan relevansi fungsionalnya, frekuensi alel minor, ketersediaan bahan referensi, dan kelayakan teknis untuk mendeteksi alel. 7 Namun, dampak tingkatan pemilihan alel ini pada frekuensi *1 dalam populasi yang berbeda belum dinilai.
Dalam studi ini, kami menghitung frekuensi alel *1 dan genotipe *1/*1 untuk 10 farmakogen dalam kelompok pemuda beragam dari Kanada Barat yang menjalani pengujian PGx menggunakan panel yang mencakup semua alel AMP Tingkat 1 dan setengah dari alel Tingkat 2. Selain itu, kami melakukan analisis in silico untuk memperkirakan bagaimana cakupan panel PGx yang diperluas akan memengaruhi frekuensi genotipe *1/*1 di seluruh kelompok leluhur. Kami berhipotesis bahwa populasi non-Eropa akan menunjukkan frekuensi alel *1 dan genotipe *1/*1 yang lebih tinggi untuk 10 farmakogen dibandingkan dengan orang Eropa, tetapi perbedaan frekuensi ini akan berkurang seiring dengan meningkatnya cakupan alel panel.
METODE
Peserta studi
Studi ini menganalisis data dari studi Pharmacogenetic-Supported Prescribing in Kids (PGx-SParK), uji coba yang mengevaluasi resep yang dipandu farmakogenetik untuk perawatan psikiatri pada remaja dan dewasa muda berusia 6–24 tahun di Kanada Barat (NCT04797364). Peserta yang memenuhi syarat berada di bawah perawatan dokter, memiliki rencana untuk memulai atau mengganti pengobatan psikiatri, dan dokter mereka bersedia menggunakan pengujian PGx untuk menginformasikan pengambilan keputusan. Individu dengan riwayat transplantasi sumsum tulang atau hati tidak diikutsertakan. Persetujuan tertulis atau persetujuan dengan persetujuan orang tua diperoleh sebelum berpartisipasi. Semua prosedur dilakukan sesuai dengan prinsip-prinsip yang dinyatakan dalam Deklarasi Helsinki dan telah disetujui oleh Dewan Etika Penelitian Kesehatan Bersama Universitas Calgary (REB20-0900).
Ukuran keturunan
Aplikasi Research Electronic Data Capture (REDCap) digunakan untuk mengumpulkan leluhur yang dilaporkan sendiri oleh peserta, menurut sembilan kelompok biogeografis PharmGKB. 8 Untuk tujuan analitis, kelompok-kelompok ini dikotomi menjadi Eropa (EUR) dan Non-Eropa (NON-EUR). Kelompok NON-EUR, yang meliputi Afrika Amerika/Afro-Karibia, Asia Timur, Asia Selatan, Latin, Afrika Sub-Sahara, Timur Dekat, Oseania, dan leluhur lain yang dilaporkan, dikonsolidasikan ke dalam satu kategori karena ukuran sampel yang kecil (<50) di beberapa kelompok biogeografis. Pendekatan ini memastikan estimasi frekuensi yang lebih stabil dan menghindari tantangan analitis yang terkait dengan frekuensi alel yang sangat rendah atau tinggi dalam subkelompok yang kurang terwakili.
Pengujian farmakogenetik dan genotipe
Sampel saliva dikumpulkan menggunakan kit Oragene OG-600 (DNA Genotek, Ottawa, Kanada), dan DNA genom diekstraksi mengikuti protokol pabrik. Genotyping dilakukan menggunakan VeriDose Core Panel v1.0 yang disesuaikan (Agena Bioscience, San Diego, AS), yang menargetkan 10 farmakogen: CYP2B6 , CYP2C19 , CYP2C9 , CYP2D6 , CYP3A4 , CYP3A5 , NUDT15 , SLCO1B1 , TPMT , dan VKORC1 . Panel tersebut mencakup semua alel Tier 1 dan 53% alel Tier 2 yang direkomendasikan oleh AMP PGx Working Group. Semua alel yang diuji dan yang belum diuji ditunjukkan pada Tabel S1 .
Analisa
Untuk semua 10 farmakogen yang diuji, frekuensi alel *1 dan genotipe *1/*1 dihitung dan dibandingkan antara kelompok EUR dan NON-EUR. Untuk menentukan kemungkinan membawa alel *1 atau genotipe *1/*1 berdasarkan garis keturunan, model regresi logistik digunakan dengan usia dan jenis kelamin sebagai kovariat. Kelompok EUR ditetapkan sebagai referensi.
Proporsi peserta kami yang salah menetapkan genotipe *1/*1 (yaitu, negatif palsu) untuk masing-masing dari 10 farmakogen diperkirakan menggunakan analisis in silico yang memperluas panel PGx kami untuk menyertakan tiga kelompok alel yang tidak diuji ( Tabel S1 ). Kelompok alel pertama memperluas panel PGx kami untuk menyertakan semua alel AMP Tier 2 yang tidak diuji. Kelompok kedua lebih lanjut memperluas panel untuk menyertakan semua alel yang tidak diuji yang dikatalogkan dalam PharmVar dengan fungsi yang meningkat, menurun, atau tidak ada. Akhirnya, kelompok ketiga memperluas panel untuk menyertakan semua alel non-normal yang tidak diuji yang dikatalogkan dalam PharmVar (fungsi meningkat, menurun, tidak ada, tidak pasti, tidak diketahui). Alel fungsi normal dalam PharmVar dikecualikan karena secara fenotip akan setara dengan *1. Pendekatan ini mengasumsikan bahwa individu yang membawa alel yang tidak diuji akan salah diklasifikasikan sebagai *1/*1 jika alel tersebut tidak disertakan dalam panel PGx. Untuk setiap alel yang belum diuji, data frekuensi untuk setiap kelompok leluhur diperoleh dari UK Biobank. 8 Jika frekuensi alel tidak tersedia di UK Biobank, kami menggunakan frekuensi yang disediakan oleh PharmGKB. 9 Untuk memperkirakan frekuensi negatif palsu, frekuensi alel yang dijumlahkan yang belum diuji dalam setiap gen digunakan untuk memperkirakan proporsi peserta yang mungkin membawa alel non-normal tetapi yang diberi genotipe *1/*1 karena keterbatasan panel. Untuk kategori NON-EUR, frekuensi negatif palsu dari masing-masing kelompok leluhur dirata-ratakan untuk menghasilkan satu perkiraan.
HASIL
Dari 1191 peserta yang terdaftar dalam studi PGx-SParK pada saat analisis ini, 105 dikecualikan karena data genotipe yang hilang ( n = 2) atau informasi leluhur ( n = 103), sehingga menyisakan sampel akhir sebanyak 1086 peserta. Di antara ini, 727 (66,9%) diidentifikasi sebagai EUR, dan 359 (33,1%) dikategorikan sebagai NON-EUR. Kelompok NON-EUR terdiri dari Afrika Amerika/Afro-Karibia ( n = 14), Amerika ( n = 46), Asia Timur ( n = 42), Latino ( n = 10), Timur Dekat ( n = 16), Oseania ( n = 1), Asia Selatan ( n = 25), Afrika Sub-Sahara ( n = 7), dan leluhur lainnya/campuran ( n = 192).
Frekuensi alel *1 dan genotipe *1/*1
Frekuensi alel *1 dan genotipe *1/*1 berbeda secara signifikan antara kelompok EUR dan NON-EUR untuk beberapa gen ( Gambar 1 ). Dibandingkan dengan partisipan EUR, partisipan NON-EUR menunjukkan frekuensi alel *1 yang lebih tinggi untuk CYP2C9 , CYP2D6 , dan CYP3A5 dan frekuensi genotipe *1/*1 yang lebih tinggi untuk CYP2C9 dan CYP3A4 . Sebaliknya, partisipan EUR menunjukkan frekuensi alel *1 yang lebih tinggi untuk CYP2C19 dan VKORC1 dan frekuensi genotipe *1/*1 yang lebih tinggi untuk VKORC1 . Frekuensi alel *1 dan genotipe *1/*1 di seluruh kelompok leluhur individu dapat ditemukan di Tabel S2 .
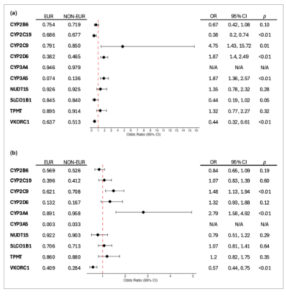
Gambar 1
Buka di penampil gambar
Kekuatan Gambar
Plot hutan dengan rasio peluang dan interval kepercayaan 95% untuk penugasan alel ( a ) *1 dan genotipe ( b ) *1/*1 pada orang non-Eropa dibandingkan dengan orang Eropa. Rasio peluang untuk CYP3A4 dan CYP3A5 tidak dilaporkan karena beberapa subkelompok non-Eropa memiliki frekuensi CYP3A4*1 100% atau frekuensi CYP3A5 *1/*1 0%, yang menyebabkan interval kepercayaan 95% berkisar dari 0 hingga tak terhingga, sehingga estimasi tidak informatif.
Perkiraan negatif palsu
Perluasan panel PGx in silico kami untuk mencakup semua alel AMP Tier 2 yang belum diuji menunjukkan perkiraan proporsi negatif palsu yang dihasilkan oleh panel kami saat ini rendah (< 1%) untuk sebagian besar gen, kecuali untuk CYP2C19 (hingga 1,8%) ( Tabel 1 ). Perluasan panel kami untuk mencakup semua alel yang belum diuji tanpa, menurun, atau meningkat fungsi di PharmVar menunjukkan bahwa perkiraan proporsi negatif palsu melebihi 1% untuk beberapa gen, termasuk SLCO1B1 (22,5%), CYP2B6 (6,0%), CYP2D6 (3,3%), CYP2C19 (2,0%), dan CYP2C9 (1,1%). Perluasan lebih lanjut untuk mencakup semua alel yang belum diuji dengan fungsi non-normal (tidak ada, menurun, meningkat, tidak pasti, tidak diketahui) dalam PharmVar menghasilkan hasil yang serupa, dengan estimasi negatif palsu yang meningkat diamati untuk SLCO1B1 (22,9%), CYP2B6 (9,7%), CYP2D6 (7,6%), CYP2C19 (2,6%), dan CYP2C9 (1,4%).
Tabel 1. Estimasi proporsi hasil negatif palsu setelah perluasan cakupan alel panel secara in silico
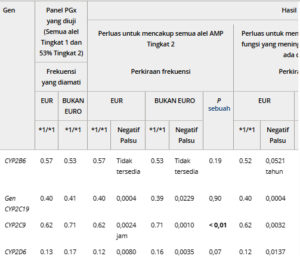
T/A menunjukkan tidak ada data frekuensi alel yang tersedia untuk memperkirakan frekuensi negatif palsu. EUR, keturunan Eropa; NON-EUR, keturunan non-Eropa. Nilai yang dicetak tebal signifikan secara statistik.
Nilai – P menunjukkan kemungkinan bahwa frekuensi *1/*1 berbeda antara kelompok. Tidak ditemukan perbedaan statistik untuk rasio negatif palsu antara kelompok EUR dan NON-EUR.
Pemeriksaan yang lebih cermat terhadap kontribusi tingkat alel menunjukkan bahwa estimasi negatif palsu yang lebih tinggi untuk SLCO1B1 terutama didorong oleh *15 (tidak berfungsi), *14 (meningkatkan fungsi), *20 (meningkatkan fungsi), *27 (fungsi tidak pasti), dan *31 (tidak berfungsi). Di antara ini, SLCO1B1 *15 menunjukkan dampak keseluruhan tertinggi pada estimasi negatif palsu, dengan 9,9% di Eropa, 8,9% di Asia Timur, dan 7,3% di populasi lain. *14 juga umum di Eropa (7,9%), sementara *20 dan *31 lebih sering terjadi pada individu keturunan Afrika (masing-masing hingga 6,0% dan 3,9%). Kontribusi tambahan berasal dari *27, yang mencapai 5,3% pada populasi Afrika Amerika/Afro-Karibia. Untuk CYP2B6 , kontributor utama meliputi *6 (fungsi berkurang) dan *7 (fungsi berkurang), dengan *6 teramati hingga 4,5% pada populasi Asia Tengah/Selatan, 4,3% pada populasi Afrika Sub-Sahara, dan 3,5% pada populasi Afrika Amerika/Afro-Karibia. Alel *7 terdapat pada 3,0% orang Asia Tengah/Selatan, dan *22 terdapat pada 3,0% individu Afrika Amerika/Afro-Karibia. Untuk CYP2D6 , CYP2C19 , dan CYP2C9 , hasil negatif palsu yang meningkat disebabkan oleh kombinasi beberapa alel, yang masing-masing memiliki frekuensi yang relatif rendah di seluruh populasi.
Pemeriksaan berdasarkan kelompok keturunan mengungkapkan bahwa frekuensi negatif palsu maksimum yang diestimasikan secara numerik, tetapi tidak secara statistik, lebih tinggi pada kelompok NON-EUR dibandingkan dengan kelompok EUR untuk semua gen ( Tabel 1 , Tabel S3 ).
DISKUSI
Temuan kami sebagian mendukung gagasan bahwa kelompok NON-EUR lebih mungkin diberi alel *1 atau genotipe *1/*1 untuk beberapa farmakogen, dan untuk sebagian individu ini, penugasan ini kemungkinan besar merupakan hasil negatif palsu. Namun, untuk farmakogen lain, hasil kami menunjukkan frekuensi alel *1 dan genotipe *1/*1 lebih tinggi pada kelompok EUR, dengan estimasi hasil negatif palsu serupa dengan yang diestimasikan pada kelompok NON-EUR. Temuan ini menyoroti bahwa kekurangan dalam cakupan alel panel memiliki implikasi potensial untuk semua populasi, dan perluasan cakupan alel kemungkinan akan mengurangi hasil negatif palsu pada semua kelompok leluhur.
Kepatuhan terhadap pedoman pemilihan alel AMP7 dianggap sebagai strategi yang efektif untuk mengurangi hasil negatif palsu. Sementara alel Tingkat 1 dianggap sebagai set minimum, temuan kami menunjukkan bahwa kegagalan untuk menyertakan semua alel Tingkat 1 dan 2 pada panel PGx dapat menghasilkan hasil yang tidak diinginkan di area terapi tertentu. Panel kami menyertakan semua alel Tingkat 1 dan sekitar setengah dari alel Tingkat 2 untuk gen yang kami uji. Namun, analisis in silico kami menunjukkan bahwa menyertakan semua alel Tingkat 2 untuk CYP2C19 dapat secara signifikan mengurangi (> 1%) frekuensi hasil negatif palsu untuk gen ini. Perluasan panel kami untuk mencakup semua alel yang diketahui dengan fungsi yang meningkat, menurun, atau tidak ada yang dikatalogkan dalam PharmVar dapat mengurangi frekuensi negatif-palsu hingga 22,5% untuk SLCO1B1 , 6,0% untuk CYP2B6 , 3,3% untuk CYP2D6 , 2,0% untuk CYP2C19 , dan 1,1% untuk CYP2C9 . Pengurangan serupa diamati ketika memasukkan semua alel fungsi yang tidak normal (meningkat, menurun, tidak ada, tidak pasti, tidak diketahui), dengan dampak terbesar terlihat pada SLCO1B1 dan CYP2B6 . Kami memperkirakan bahwa penyertaan SLCO1B1 *15 (tidak ada fungsi) dan *14 (peningkatan fungsi) dapat menurunkan tingkat negatif-palsu absolut hampir 18% pada orang Eropa dan 9% pada orang Asia Timur; SLCO1B1 *27 (fungsi tidak pasti) dan *31 (tidak berfungsi) dapat mengurangi angka hingga 9% pada kelompok keturunan Afrika. Penambahan CYP2B6*6 dan *7 (keduanya menurunkan fungsi) dapat mengurangi hasil negatif palsu hingga 4% pada orang Asia Tengah/Selatan.
Pengujian untuk semua varian yang diketahui dapat difasilitasi oleh pengurutan generasi berikutnya (bacaan pendek dan panjang) (NGS). Namun, penerapan klinis pendekatan NGS memiliki tantangan penggantian biaya, regulasi, teknis, dan etika yang signifikan yang perlu ditangani sebelum adopsi rutin untuk pengujian PGx. 10 , 11 Demikian pula, banyak varian yang diketahui memiliki fungsi yang tidak diketahui atau tidak pasti. Misalnya, 59% (99/168) dan 40% (19/48) dari varian CYP2D6 dan CYP2B6 yang diketahui yang dikatalogkan dalam PharmVar, masing-masing, memiliki fungsi yang tidak diketahui, tidak pasti, atau tidak ditetapkan. Akibatnya, pengujian varian ini akan mengurangi frekuensi alel *1 tetapi akan meningkatkan jumlah temuan yang tidak pasti dan mempersulit penerapan klinis. Dengan demikian, pekerjaan tambahan diperlukan untuk mengkarakterisasi varian ini sebelum memasukkannya pada panel pengujian PGx. Sementara itu, strategi yang masuk akal untuk mengurangi hasil negatif palsu, berdasarkan temuan kami, adalah dengan mengadopsi panel PGx yang mencakup semua alel AMP Tier 1 dan 2 untuk gen yang diuji. Untuk gen tanpa pedoman AMP (misalnya, CYP2B6 , SLCO1B1 ), set alel optimal belum ditentukan, tetapi hasil kami menunjukkan bahwa penyertaan SLCO1B1 *5, *14, *15, dan *31 serta CYP2B6 *4, *6, *7, *9, dan *18 akan mengurangi hasil negatif palsu. Namun, efektivitas biaya dari perluasan cakupan panel juga harus dipertimbangkan, karena pengujian yang lebih luas dapat meningkatkan akurasi tetapi meningkatkan biaya implementasi klinis.
Beberapa peringatan dari penelitian ini harus dipertimbangkan ketika menafsirkan hasilnya. Pertama, tidak ada frekuensi ‘kebenaran’ atau referensi yang tersedia untuk *1/*1 di semua leluhur. Dengan demikian, tidak jelas berapa proporsi setiap kelompok leluhur yang benar-benar tidak memiliki varian non-normal untuk setiap farmakogen yang kami periksa. Kami berasumsi semua individu yang diberi genotipe *1/*1 berpotensi negatif palsu, tetapi ada kemungkinan beberapa dari individu ini adalah negatif sejati atau membawa varian yang memiliki fungsi normal, yang tidak akan menghasilkan penunjukan negatif palsu. Dengan demikian, estimasi kami mewakili frekuensi negatif palsu maksimum yang mungkin berdasarkan basis pengetahuan saat ini. Selain itu, dampak fungsional dari banyak alel yang diklasifikasikan sebagai ‘fungsi non-normal’ masih belum sepenuhnya dicirikan, yang menambah ketidakpastian pada klasifikasi dan relevansi klinis potensial mereka. Kedua, kami menggunakan analisis in silico untuk memperkirakan frekuensi negatif palsu. Sebaiknya, sampel peserta akan diuji pada serangkaian panel (misalnya, hanya alel Tier 1, Tier 1 dan 2, panel NGS) dan kemudian dibandingkan secara berdampingan untuk mendapatkan frekuensi negatif palsu yang sebenarnya. Ketiga, leluhur ditentukan oleh laporan diri. Idealnya, ini akan ditetapkan menggunakan pendekatan genomik, tetapi data genomik yang diperlukan tidak tersedia. Namun, dapat dikatakan bahwa leluhur laporan diri lebih relevan secara klinis daripada leluhur genomik karena yang terakhir jarang tersedia secara klinis. Akhirnya, ada bias inheren dalam sumber data frekuensi alel seperti UK Biobank dan PharmGKB, yang sebagian besar mencakup individu dari kelompok EUR. Bias ini menyebabkan kesenjangan data dan memengaruhi estimasi kuat frekuensi negatif palsu untuk pharamcogenes di beberapa kelompok NON-EUR. Akibatnya, frekuensi negatif palsu dalam kelompok NON-EUR mungkin bias terhadap kelompok leluhur dengan data frekuensi alel phamakogen yang lebih banyak tersedia serta kelompok dengan representasi yang lebih besar dalam kumpulan data kami. Selain itu, karena kelompok studi kami terdiri dari individu yang menjalani perawatan psikiatris, frekuensi alel dan genotipe mungkin tidak sepenuhnya mewakili populasi umum, sehingga menimbulkan potensi bias seleksi.
Singkatnya, peluang untuk mendapatkan alel *1 dan genotipe *1/*1 bervariasi menurut kelompok leluhur dan farmakogenetik, dengan estimasi frekuensi negatif palsu di atas 1% untuk beberapa farmakogenetik dalam kelompok EUR dan NON-EUR. Bertentangan dengan hipotesis kami, partisipan NON-EUR tidak memiliki frekuensi alel *1 dan genotipe *1/*1 yang lebih tinggi untuk semua farmakogenetik yang kami uji. Dengan demikian, perluasan cakupan panel PGx kemungkinan akan mengurangi risiko negatif palsu terlepas dari populasi yang menjalani pengujian farmakogenetik.

