
Abstrak
SARS-CoV-2, virus yang bertanggung jawab atas pandemi COVID-19, telah mengalami evolusi berkelanjutan, yang mengarah pada munculnya varian dengan penularan yang berubah dan penghindaran imun. Untuk protein non-struktural (Nsps) SARS-CoV-2, terdapat analisis struktural terbatas dari mutasi yang terjadi secara alami. Di sini, kami mengidentifikasi empat polimorfisme nukleotida tunggal non-sinonim (nsSNPs) dalam garis keturunan Epsilon SARS-CoV-2 dalam Nsp15, endoribonuklease yang penting untuk penghindaran imun. Dari nsSNP Epsilon ini, E266Q berada dalam domain katalitik. Studi ini menyelidiki efeknya pada aktivitas enzimatik, stabilitas struktural, dan perakitan oligomerik dengan kristalografi serial. Dengan memecahkan struktur heksamer Nsp15 pada suhu kamar dari Nsp15-E266Q dan WT dalam grup ruang P2 1 hingga 3 Å, kami mengamati gerakan asimetris dalam subunit trimernya, suatu fitur yang tidak terlihat dalam grup ruang simetri tinggi yang dilaporkan sebelumnya. Gerakan asimetris ini menyerupai perubahan konformasi yang diinduksi substrat yang dilaporkan dalam struktur Nsp15 terikat RNA, yang menunjukkan relevansi fungsional. Analisis biokimia lebih lanjut mengungkapkan bahwa Nsp15-E266Q menunjukkan aktivitas enzimatik dan stabilitas termal yang jauh lebih tinggi dibandingkan dengan protein tipe liar. Temuan ini menyoroti bagaimana mutasi pada Nsp15 berkontribusi pada replikasi virus dan penghindaran imun, menawarkan wawasan tentang mekanisme molekuler yang mendasari evolusi varian SARS-CoV-2 dan strategi terapi potensial.
1. PENDAHULUAN
Severe Acute Respiratory Syndrome Coronavirus 2 (SARS-CoV-2) bertanggung jawab atas pandemi COVID-19, yang menyebabkan morbiditas dan mortalitas global yang signifikan sejak kemunculannya pada bulan Desember 2019 (Machhi et al. 2020 ). SARS-CoV-2 adalah virus RNA berselubung, positif-sense dengan genom ~30 kb yang tidak tersegmentasi (Wu et al. 2020 ). Genom mengkodekan empat protein struktural, beberapa protein aksesori, dan dua poliprotein besar (Pp1a dan Pp1ab), yang diproses pasca-translasi menjadi 16 protein non-struktural (Nsps). Nsps berkumpul menjadi kompleks replikase-transkriptase (RTC) besar yang melakukan aktivitas enzimatik dan pengikatan RNA penting yang diperlukan untuk replikasi dan infeksi virus (Galanopoulos et al. 2020 ; Kim et al. 2020a ). Urutan dan fungsi Nsp ini sangat terpelihara di antara virus corona (Kim et al. 2020b ), menjadikannya target yang menarik untuk pengembangan obat antivirus.
Genom SARS-CoV-2 terus berevolusi, mengakumulasi mutasi yang menyebabkan munculnya varian yang lebih patogen. Organisasi Kesehatan Dunia mengkategorikan varian ini berdasarkan kemampuan penularan dan penghindaran imunnya menjadi varian yang perlu diwaspadai (VOC) atau varian yang menarik (VOI). VOC yang terkenal termasuk Alpha (B.1.1.7), Beta (B.1.351), Gamma (P.1), Epsilon (B.1.427, B.1.429), Delta (B.1.617.2), dan Omicron (B.1.1.529) (Tabel S1 , Informasi Pendukung) yang masing-masing varian barunya menunjukkan peningkatan kemampuan penularan dan patogenisitas daripada SARS-CoV-2 asli, yang menyebabkan jutaan kematian di seluruh dunia (CDC 2023 ). VOI seperti Lambda dan Mu telah menyebabkan dampak yang kurang signifikan. Per September 2023, Pusat Pengendalian dan Pencegahan Penyakit AS (CDC) mencantumkan garis keturunan induk Omicron (BA.1 atau serupa) sebagai VOC, sementara varian lain seperti Alpha, Beta, Gamma, Epsilon, Kappa, Lambda, Eta, Mu, Iota, dan Zeta diklasifikasikan sebagai Varian yang Sedang Dipantau (VBM) (CDC 2023 ).
Varian Epsilon, pertama kali terdeteksi di California, pada bulan Juli 2020, menunjukkan peningkatan penularan dibandingkan dengan varian lokal yang lazim pada saat itu. Varian Epsilon awalnya dikenali sebagai VOC oleh CDC. Dengan munculnya varian lain, dampak Epsilon menurun seiring waktu. Epsilon dikenali sebagai VOI pada bulan Februari 2021 dan kemudian diubah menjadi VBM pada bulan September 2021 (CDC 2023 ). Protein lonjakan Epsilon mengandung empat polimorfisme nukleotida tunggal non-sinonim (nsSNP) yang terkenal, S13I, W152C, D614G, dan L452R, dengan D614G sedang dipelajari secara ekstensif dalam konteks peningkatan masuknya virus (Zhang et al. 2021 ). NsSNP dalam protein non-struktural dari garis keturunan Epsilon masih relatif belum dieksplorasi.
Protein nonstruktural Nsp15, yang juga dikenal sebagai endoribonuklease spesifik uridilat, memainkan peran penting dalam lolosnya virus dari sistem imun inang (Hackbart et al. 2020 ). Nsp15 membelah urutan poliuridin dari genom RNA negatif SARS-CoV-2, yang sebaliknya bertindak sebagai pola molekuler terkait patogen (PAMP) yang dikenali oleh reseptor pengenal pola MDA5 inang (Bhardwaj et al. 2006 ; Frazier et al. 2021 ; Hackbart et al. 2020 ). Nsp15, protein dengan residu 345 dan berat 39 kDa, terdiri dari tiga domain: domain oligomerisasi N-terminal (asam amino 1–64), domain tengah (asam amino 65–182), dan domain katalitik C-terminal (asam amino 207–347) (Kim et al. 2020b ; Pillon et al. 2021 ). Enzim ini bergantung pada Mn 2+ dan berfungsi sebagai heksamer, yang terdiri dari dimer trimer. Struktur kristal apo Nsp15 (Jernigan et al. 2023 ; Kim et al. 2020b ) dan Nsp15 dalam kompleks dengan inhibitor (Kim et al. 2021 ) dari SARS-CoV-2 sebagian besar telah dipecahkan dalam grup ruang heksagonal (P6 3 ), yang memperlihatkan heksamer yang terdiri dari dua homotrimer berdasarkan satu monomer dari setiap trimer dalam unit asimetris. Susunan heksamerik serupa telah diamati dalam struktur Nsp15 dari virus korona lain, seperti SARS-CoV (Bhardwaj et al. 2008 ; Stefano Ricagno et al. 2006 ), Middle East Respiratory Syndrome-CoV (MERS-CoV) (Zhang et al. 2018 ), dan mouse hepatitis virus (MHV) (Xu et al. 2006 ). Organisasi heksamerik ini, yang ditinjau dalam van Loy et al. ( 2024 ), menyoroti arsitektur yang dilestarikan di seluruh virus korona. Struktur suhu-ruangan Nsp15 telah menunjukkan perbedaan dalam fleksibilitas antara kedua trimer (Jernigan et al. 2023 ), sebuah pengamatan yang dikonfirmasi oleh struktur cryo-EM (Pillon et al. 2021 ). Studi cryo-EM dengan adanya substrat RNA untai tunggal (ssRNA) dan RNA untai ganda (dsRNA) (Frazier et al. 2022a ; Ito et al. 2024 ) telah mengungkapkan asimetri sederhana dalam heksamer yang disebabkan oleh pengikatan substrat. Sementara studi mutasi di beberapa varian SARS-CoV-2 telah dilakukan (Wilson et al. 2022 ), studi struktural komprehensif pada Nsp15 dari varian SARS-CoV-2 yang muncul masih terbatas.
Dalam studi ini, kami menyelidiki sifat struktural dan fungsional Nsp15 dari garis keturunan Epsilon, dengan fokus pada efek nsSNP pada fungsi enzimatik. Secara khusus, kami mengidentifikasi mutasi polimorfisme nukleotida tunggal non-sinonim (nsSNP), E266Q dalam Nsp15 dari Epsilon dan mengkarakterisasi dampaknya pada ekspresi protein, aktivitas enzimatik, dan stabilitas struktural. Lebih jauh, kami menentukan struktur Nsp15-E266Q pada suhu kamar dengan kristalografi milidetik serial. Dalam kristalografi milidetik serial, data sinar-X dikumpulkan di Synchrotrons (ESRF) pada suhu kamar dalam mode bidikan sinar-X tunggal dari jutaan mikrokristal dalam dudukan target tetap, dengan 250 bidikan sinar-X tunggal per detik. Hasilnya mengungkapkan gerakan asimetris intrinsik dalam rakitan heksamerik, yang memberikan wawasan tentang perubahan struktural dan dampak fungsional dari mutasi ini. Berdasarkan pengamatan ini, kami menganalisis ulang data kristalografi tipe liar (WT) dari penelitian sebelumnya (Jernigan et al. 2023 ), dan meskipun kurang jelas, menemukan gerakan asimetris yang sama dalam protein WT Nsp15.
2 HASIL
2.1 Identifikasi SNP non-sinonim di Nsp15 dalam garis keturunan Epsilon
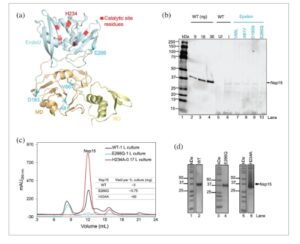
Untuk memeriksa efek struktural dan fungsional mutasi pada Nsp15 dari garis keturunan SARS-CoV-2 Epsilon, kami melakukan penyelarasan sekuens genom antara varian Epsilon dan galur asli Wuhan SARS-CoV-2. Dari penyelarasan ini, kami mengekstrak wilayah sekuens yang sesuai dengan Nsp15. Analisis ini mengidentifikasi empat polimorfisme nukleotida tunggal non-sinonim (nsSNP) yang mengarah pada substitusi asam amino: V66L, A81V, D183N, dan E266Q. Pemetaan struktural mutasi ini (Gambar 1a ) mengungkapkan bahwa V66L, A81V, dan D183N terletak di dalam domain tengah, sementara E266Q terletak di dalam domain katalitik dan sebelumnya terlibat dalam interaksi langsung dengan monomer yang berdekatan (Bhardwaj et al. 2008 ).
GAMBAR 1
Buka di penampil gambar
Presentasi PowerPoint
Lokalisasi struktural nsSNP Epsilon Nsp15 dan ekspresi serta pemurnian nsSNP Nsp15 dibandingkan dengan WT. (a) Representasi struktural monomer Nsp15 (entri PDB 7K9P , rantai A; Jernigan et al. 2023 ) yang menyoroti posisi nsSNP yang diidentifikasi dalam garis keturunan Epsilon. Residu yang diubah oleh nsSNP ditunjukkan dengan warna biru, dengan residu katalitik yang ditandai. Residu situs katalitik H234A diberi label khusus. (b) Analisis imunoblot Anti-His dari lisat sel utuh yang mengekspresikan WT dan nsSNP dari garis keturunan Epsilon. Jalur 1: tangga berat molekul protein; Jalur 2–4: WT murni yang dimuat pada 9, 18, dan 36 ng, masing-masing; Jalur 5: lisat sel utuh WT yang tidak diinduksi; Jalur 6: Lisat sel utuh yang diinduksi IPTG dari WT; Jalur 7–10: Lisat sel utuh yang diinduksi IPTG yang mengandung nsSNP garis keturunan Epsilon. (c) Profil kromatografi pengecualian ukuran (SEC) yang membandingkan elusi protein dari WT (hitam, kultur 1 L), Nsp15-E266Q (biru, kultur 1 L), dan H234A (merah, kultur 0,17 L). Hasil protein (mg per kultur 1 L) ditunjukkan. (d) Gel SDS-PAGE yang diwarnai Coomassie dari Nsp15 yang dimurnikan sesuai dengan puncak SEC pada (c), yang mengonfirmasi kemurnian protein. Jalur 1, 3, 5: tangga berat molekul protein; Jalur 2: WT; Jalur 4: Nsp15-E266Q; Jalur 6: H234A.
Domain katalitik Nsp15 sangat penting untuk aktivitas endoribonuklease-nya, yang memfasilitasi penghindaran imun dengan memecah urutan poliuridin dalam RNA virus (Frazier et al. 2021 ; Hackbart et al. 2020 ). Kehadiran E266Q dalam domain ini menunjukkan potensi perubahan aktivitas enzimatik akibat perubahan struktur lokal atau gangguan interaksi yang penting untuk katalisis.
2.2 Studi ekspresi Nsp15-E266Q menunjukkan aktivitas penghambatan autoimun
Untuk mengevaluasi ekspresi semua nsSNP dari Epsilon, kami menggunakan Escherichia coli untuk produksi protein rekombinan. Tingkat ekspresi dianalisis dari lisat sel utuh dan divisualisasikan dengan Western blot anti-His. Protein WT yang dimurnikan dari SARS-CoV-2 asli dimuat dalam tiga jumlah berbeda (9 ng, 18 ng, 36 ng) sebagai referensi (Gambar 1b , lajur 2–4). Kami mengamati bahwa Nsp15-E266Q menunjukkan tingkat ekspresi yang secara signifikan lebih rendah (Gambar 1b , lajur 10) dibandingkan dengan tipe liar (WT) (Gambar 1b , lajur 6), seperti yang ditunjukkan oleh pita lemah pada Western blot anti-His.
Dengan mempertimbangkan ekspresi Nsp15-E266Q yang berkurang dibandingkan dengan WT, kami melakukan analisis komparatif terhadap hasil pemurnian protein. Untuk memungkinkan perbandingan ini, kami menyertakan mutan H234A yang tidak aktif secara katalitik, yang sebelumnya telah dikarakterisasi baik dalam SARS-CoV (Guarino et al. 2005 ) dan SARS-CoV-2 (Guarino et al. 2005 ; Otter et al. 2024 ). Mutasi situs aktif ini memungkinkan Nsp15 untuk mengikat substrat RNA sambil menghilangkan kemampuannya untuk membelahnya, menjadikannya kontrol yang berharga untuk eksperimen yang melibatkan Nsp15-E266Q dan WT. Khususnya, penelitian sebelumnya telah menunjukkan bahwa H234A diekspresikan pada tingkat yang lebih tinggi daripada WT baik dalam SARS-CoV (Guarino et al. 2005 ) dan SARS-CoV-2 (Otter et al. 2024 ). Konsisten dengan laporan ini, data kami menunjukkan bahwa H234A menunjukkan jumlah heksamer Nsp15 murni yang sangat tinggi. Setelah kromatografi afinitas Ni-NTA, profil kromatografi pengecualian ukuran (SEC) menunjukkan hasil pemurnian sekitar 80 mg per liter kultur untuk H234A, dibandingkan dengan ~3 mg per liter untuk WT dan 0,75 mg untuk Nsp15-E266Q, sebagaimana diukur menggunakan spektrofotometer Nanodrop (Gambar 1c ). Analisis SDS-PAGE dari puncak SEC dari Nsp15 murni (Gambar 1c ) menunjukkan pita berbeda yang sesuai dengan berat molekul Nsp15 yang diharapkan (42 kDa) untuk ketiga protein (Gambar 1d ). Analisis lebih lanjut terhadap fraksi SEC dari WT dan Nsp15-E266Q mengungkapkan distribusi protein yang serupa di seluruh puncak kromatografi (Gambar S1a–d ), yang menunjukkan bahwa mutasi E266Q tidak mengubah status oligomerik Nsp15 secara signifikan. Pengamatan ini menunjukkan bahwa meskipun Nsp15-E266Q dapat diekspresikan dan dimurnikan, hasil rekombinannya secara signifikan lebih rendah daripada WT dan mutan H234A.
2.3 Nsp15-E266Q menunjukkan peningkatan aktivitas katalitik dibandingkan dengan tipe liar
Mengingat bahwa mutasi E266Q terletak dalam domain katalitik Nsp15, kami berhipotesis bahwa nsSNP ini dapat memengaruhi aktivitas katalitik enzim. Untuk menyelidiki ini, kami menggunakan uji yang telah ditetapkan sebelumnya yang digunakan untuk evaluasi aktivitas WT (entri PDB: 7K9P ) menggunakan substrat RNA 21-nukleotida, (Jernigan et al. 2023 ) yang diberi label pada ujung 3′ dengan fluorofor (Gambar 2a ). Untuk menguji apakah mutasi E266Q secara drastis memengaruhi aktivitas katalitik Nsp15, kami melakukan uji in vitro. Baik Nsp15-E266Q dan WT dapat membelah substrat RNA, menghasilkan fragmen dengan ukuran yang diharapkan (Gambar 2b , lajur 3 dan 4). Seperti yang diharapkan, mutan H234A yang tidak aktif secara katalitik dan kontrol buffer tidak menunjukkan aktivitas pembelahan yang terdeteksi (Gambar 2b , jalur 1 dan 2).

GAMBAR 2
Buka di penampil gambar
Presentasi PowerPoint
Aktivitas fungsional, kinetika enzimatik, dan stabilitas termal varian Nsp15. (a) Substrat RNA 21 nukleotida (nt) yang diberi label dengan fluorofor NIR-800 (Near Infra-Red 800 nm) pada ujung 3′. Segitiga merah menunjukkan situs pembelahan antara U dan G, menghasilkan produk 18 nt. (b) Uji fungsional berbasis gel Urea-PAGE yang dilakukan menggunakan substrat RNA yang ditunjukkan pada (a). Jalur 1: buffer SEC (tanpa kontrol enzim); Jalur 2: mutan H234A yang tidak aktif secara katalitik; Jalur 3: WT; Jalur 4: Nsp15-E266Q. (c) Uji kinetik berbasis fluoresensi yang dilakukan dengan substrat RNA fluorogenik 6-FAM-5′dArUdAdA3′-6-TAMRA. Reaksi kontrol tanpa enzim ditunjukkan dalam garis putus-putus. (d) Plot Michaelis–Menten untuk WT dan Nsp15-E266Q, yang menunjukkan perbedaan dalam parameter kinetik ( V max dan K m ). Kesalahan menunjukkan deviasi standar dari rangkap tiga. (e) Analisis stabilitas termal WT, Nsp15-E266Q , dan H234A menggunakan plot turunan pertama dari fluoresensi relatif versus suhu. Suhu transisi untuk tahap pelipatan termal pertama ( T m ke-1 ) dan kedua ( T m ke-2 ) ditunjukkan.
Untuk mengukur dan mengkarakterisasi perilaku katalitik Nsp15-E266Q lebih lanjut, kami melakukan uji kinetik berbasis fluoresensi menggunakan oligonukleotida RNA fluorogenik, 6-FAM-5′dArUdAdA3′-6-TAMRA. Substrat ini mengandung fluorofor (6-karboksifluorescein, FAM) pada ujung 5′ dan peredam (6-karboksitetrametilrodamin, TAMRA) pada ujung 3′, yang memungkinkan presisi dan sensitivitas tinggi dalam pemantauan sintesis produk. Uji ini mengungkapkan bahwa Nsp15-E266Q menunjukkan peningkatan aktivitas dibandingkan dengan WT (Gambar 2c ). Selain itu, parameter kinetik ditentukan, dengan kecepatan maksimum ( V max ) dan konstanta Michaelis ( K m ) dihitung pada 8720 RFU/menit dan 1,37 μM untuk Nsp15-E266Q dan 6144,6 RFU/menit dan 1,57 μM untuk WT (Gambar 2d ).
2.4 Nsp15-E266Q menunjukkan peningkatan stabilitas termal dibandingkan dengan tipe liar
Untuk menyelidiki apakah perbedaan yang diamati dalam aktivitas katalitik terkait dengan perubahan dalam stabilitas struktural Nsp15, kami melakukan fluorimetri pemindaian diferensial (Gambar 2e ). Nsp15-E266Q, H234A, dan WT dianalisis pada rentang suhu 30–95 °C. Konsisten dengan penelitian sebelumnya pada WT (Choi et al. 2021 ; Kim et al. 2020b ), dua suhu transisi termal yang berbeda ( Tm ) diamati: transisi awal pada sekitar 45 °C, kemungkinan sesuai dengan disosiasi heksamer (Kim et al. 2020b ), dan transisi kedua pada sekitar 58 °C, sesuai dengan pelipatan monomer (Kim et al. 2020b ). Menariknya, Nsp15-E266Q menunjukkan sedikit peningkatan dalam stabilitas termal dibandingkan dengan WT dan H234A, dengan nilai Tm untuk transisi pertama ( Tm ke – 1 ) dan kedua ( Tm ke-2) diukur pada 46,5 dan 60,5°C, berturut – turut. Sebaliknya, WT dan H234A menunjukkan nilai Tm sekitar 44–46° C dan 58,5°C untuk transisi pertama dan kedua. Hasil ini menunjukkan bahwa mutasi E266Q meningkatkan termostabilitas Nsp15 relatif terhadap WT dan mutan H234A yang tidak aktif secara katalitik.
Struktur kristal 2,5 Nsp15-E266Q dalam grup ruang P6 3
Untuk menyelidiki perubahan struktural potensial yang disebabkan oleh mutasi E266Q, yang dapat menjelaskan peningkatan aktivitas enzimatik dan stabilitas termalnya, kami menentukan struktur kristal Nsp15-E266Q menggunakan kristalografi sinar-X serial. Nsp15-E266Q yang dimurnikan dikristalkan menggunakan metode kristalisasi batch, menghasilkan kristal seperti jarum dengan panjang sekitar 50–100 μm (Gambar S2 a ). Lima mikroliter suspensi kristal dimuat pada penahan target tetap menggunakan sistem pengiriman target tetap foil-on-foil. Data dikumpulkan pada garis sinar ID-29 di Fasilitas Radiasi Sinkrotron Eropa (ESRF). Pengumpulan data sinar-X dilakukan dalam mode bidikan tunggal pada suhu kamar di mana data difraksi dari kristal dikumpulkan dengan 250 bidikan sinar-X sepanjang 90 μs/detik menggunakan chopper. Chip secara otomatis dipindahkan sejauh 50 μm di antara bidikan sinar-X. Pengumpulan data sebanyak 40.000 gambar/chip dilakukan dalam waktu 8 menit/chip (Doak et al. 2024 ). Set data terdiri dari 54.091 gambar yang diindeks dan strukturnya diselesaikan dengan resolusi 3 Å (Tabel 1 dan Gambar S2 b ).
TABEL 1. Statistik pemrosesan dan penyempurnaan data.

Nsp15 diketahui membentuk heksamer yang terdiri dari dua homotrimer, dengan perbedaan fleksibilitas antara trimer (Jernigan et al. 2023 ). Karena studi kristalografi Nsp15 sebelumnya dilakukan dalam grup ruang P6 3 dengan setiap unit asimetris mengandung satu monomer dari setiap trimer (Jernigan et al. 2023 ; Kim et al. 2020b ; Kim et al. 2021 ), kami awalnya memecahkan struktur Nsp15-E266Q dalam grup ruang ini (Gambar S2 b ). Perbandingan dengan studi sebelumnya tentang struktur WT suhu ruangan, yang dipecahkan dengan kristalografi sinar-X serial menggunakan laser elektron bebas sinar-X (XFEL) (entri PDB 7K9P ; Jernigan et al. 2023 ), menunjukkan kesamaan struktural yang tinggi, dengan deviasi akar rata-rata kuadrat (RMSD) untuk Cα sebesar 0,98 Å. Meskipun konfigurasi keseluruhannya kekal, Nsp15-E266Q (Gambar 3 , kanan) menunjukkan faktor B keseluruhan yang lebih tinggi dibandingkan dengan WT (Gambar 3 , kiri), yang menunjukkan peningkatan fleksibilitas dalam struktur mutan.

GAMBAR 3
Buka di penampil gambar
Presentasi PowerPoint
Perbandingan faktor-B antara Nsp15 WT (entri PDB 7K9P ) dan Nsp15-E266Q diselesaikan dalam P6 3. Representasi kartun dari struktur Nsp15 yang menunjukkan faktor-B untuk WT (entri PDB 7K9P , kiri; Jernigan et al. 2023 ) dan Nsp15-E266Q (kanan). Wilayah dengan faktor-B terendah (menunjukkan fleksibilitas paling rendah) ditunjukkan dengan warna biru, sementara wilayah dengan faktor-B tertinggi (menunjukkan fleksibilitas paling tinggi) ditunjukkan dengan warna merah. Gradien warna berkisar dari 20 Å 2 (biru tua) hingga 110 Å 2 (merah tua).
Struktur 2.6 Nsp15-E266Q dalam grup ruang P2 1 menunjukkan gerakan asimetris
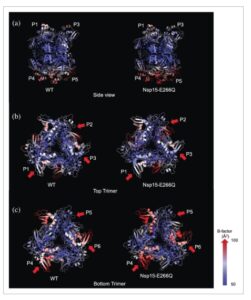
Bahasa Indonesia: Untuk lebih memahami faktor-B yang lebih tinggi yang diamati di Nsp15-E266Q dan mengeksplorasi potensi asimetri struktural, kami memecahkan strukturnya dalam grup ruang P2 1 pada resolusi 3 Å (Tabel 1 ). Sebagai perbandingan, kami juga memecahkan struktur WT (entri PDB 7K9P ) dalam grup ruang yang sama (P2 1 ). Tidak seperti struktur dalam grup ruang P6 3 , yang mengandung satu monomer dari setiap trimer dalam unit asimetris (Jernigan et al. 2023 ; Kim et al. 2020b ; Kim et al. 2021 ), grup ruang P2 1 mengandung heksamer penuh dalam unit asimetris, meminimalkan potensi bias simetri. Analisis ini mengungkapkan bahwa kedua trimer dari heksamer tersebut memang secara intrinsik asimetris dalam dirinya sendiri sebagaimana berkaitan dengan faktor-B. Konsisten dengan pengamatan sebelumnya, satu trimer menunjukkan faktor-B yang lebih tinggi secara keseluruhan, yang menunjukkan fleksibilitas yang lebih besar. Selain itu, Nsp15-E266Q memiliki faktor-B keseluruhan yang sedikit lebih tinggi dibandingkan dengan WT. Oleh karena itu, kami telah menetapkan setiap monomer label unik dengan P1, P2, dan P3 membentuk trimer yang kurang fleksibel, dan P4, P5, dan P6 membentuk trimer kedua yang lebih fleksibel (Gambar 4a ). Di dalam trimer teratas yang kurang fleksibel, satu monomer (P2) memiliki faktor-B yang lebih tinggi dibandingkan dengan dua monomer lainnya (P1 dan P3) (Gambar 4b ). Demikian pula, pada trimer yang lebih fleksibel, satu monomer (P6) menunjukkan faktor-B yang lebih tinggi dibandingkan dengan dua monomer lainnya (P4 dan P5) (Gambar 4c ). Gerakan asimetris ini dalam setiap trimer juga diidentifikasi secara independen dari studi mikroskopi kriogenik-elektron (Cryo-EM) dari Nsp15 yang terikat pada RNA (Frazier et al. 2022b ; Ito et al. 2024 ). Studi-studi yang dipublikasikan ini mengungkapkan beberapa atribut kunci dari pengikatan substrat Nsp15: Pertama, substrat berinteraksi secara asimetris dengan dua monomer dari satu trimer dan meluas untuk menghubungi monomer dari trimer kedua. Kedua, pengikatan substrat menginduksi sangat sedikit asimetri antar-monomer. Ketiga, resolusi lokal monomer, proksi untuk dinamika dalam cryo-EM, konsisten dengan pengamatan kami terhadap Nsp15-E266Q dalam grup ruang P2 1. Menariknya, tingkat gerakan asimetris dalam trimer lebih jelas pada Nsp15-E266Q dibandingkan dengan WT, menyoroti fitur struktural yang berpotensi terkait dengan peningkatan aktivitas katalitik dan stabilitas termal mutasi. Tingkat detail ini tidak dapat diamati dalam struktur kristalografi Nsp15 sebelumnya yang dipecahkan dalam grup ruang P6 3 , menyoroti pentingnya investigasi struktural pada simetri yang lebih rendah.
GAMBAR 4
Buka di penampil gambar
Presentasi PowerPoint
Struktur WT dan Nsp15-E266Q yang dipecahkan dalam grup ruang P2 1 memperlihatkan perubahan dalam gerakan asimetris. (a) Tampilan samping struktur WT (kiri) dan Nsp15-E266Q (kanan) ditunjukkan dengan warna berdasarkan faktor-B, yang menyorot variabilitas faktor-B di seluruh heksamer dan di dalam monomer yang membentuk trimer. Monomer diberi label dari P1 hingga P6. (b) Tampilan atas WT (kiri) dan Nsp15-E266Q (kanan) dari trimer atas. Perbedaan dalam daerah fleksibilitas ditunjukkan dengan panah merah. (c) Tampilan atas WT (kiri) dan Nsp15-E266Q (kanan) dari trimer bawah. Gradien warna membentang dari 50 Å 2 (biru tua) hingga 100 Å 2 (merah tua).
3 DISKUSI
Dalam studi ini, kami menyelidiki sifat fungsional dan struktural dari mutasi E266Q, mutasi pada domain katalitik Nsp15 dari garis keturunan SARS-CoV-2 Epsilon, untuk peran potensialnya dalam peningkatan virulensi. Nsp15-E266Q menunjukkan aktivitas enzimatik yang secara signifikan lebih tinggi daripada protein tipe liar (WT), seperti yang ditunjukkan oleh uji pembelahan titik akhir dan kinetika fluoresensi, yang dapat meningkatkan replikasi virus dan manipulasi sel inang, berkontribusi pada peningkatan penularan garis keturunan Epsilon. Namun, Nsp15-E266Q menunjukkan tingkat ekspresi dan hasil pemurnian yang berkurang, kemungkinan karena pembelahan mRNA-nya sendiri selama penerjemahan. Analisis stabilitas termal menunjukkan bahwa Nsp15-E266Q memiliki suhu transisi termal yang sedikit lebih tinggi ( Tm ) daripada WT, yang menunjukkan bahwa ia lebih stabil.
Untuk mengevaluasi konservasi residu E266 di seluruh virus korona, kami melakukan penyelarasan sekuens ganda Nsp15 dari SARS-CoV-2, SARS-CoV, MERS-CoV, MHV, dan HCoV-229E. Penyelarasan tersebut mengungkapkan bahwa sementara mutasi E266Q spesifik untuk garis keturunan Epsilon SARS-CoV-2, posisi ekuivalen dalam MHV mengandung glutamin (Q), yang menunjukkan potensi kesamaan fungsional antara protein-protein ini (Gambar S3 ). Pengamatan ini dapat menunjukkan peran glutamin yang dilestarikan pada posisi ini dalam virus korona tertentu, yang dapat memiliki implikasi struktural atau katalitik.
Mutasi pada domain katalitik Nsp15 sering memengaruhi aktivitas enzimatik dan stabilitas struktural, karena wilayah ini penting untuk menjaga integritas domain katalitik, lipatan EndoU (Wilson et al. 2022 ). Mutasi E266Q sendiri terletak di sisi terjauh dari salah satu lembar β antiparalel yang menyusun kantong katalitik tepat di seberang residu situs aktif K289. Studi struktural E266A sebelumnya pada SARS-CoV menunjukkan bahwa residu membuat jembatan garam antar-monomerik langsung dengan R61, yang telah terbukti penting untuk oligomerisasi (Bhardwaj et al. 2008 ). Sementara mutasi E266A mengganggu oligomerisasi pada SARS-CoV, kami tidak mengamati perubahan seperti itu pada Nsp15 SARS-CoV-2. Ini menunjukkan bahwa E266 memiliki peran rheostatik pada SARS-CoV-2. Pelemahan jembatan garam antar-monomerik antara E266 dan R61 dalam Nsp15-E266Q menyebabkan pergerakan yang lebih besar dalam domain enzimatik, yang pada gilirannya kemungkinan meningkatkan pergantian katalitik. Motilitas tambahan ini juga dapat memfasilitasi perubahan konformasi fungsional yang diperlukan untuk meningkatkan aktivitas katalitik. Oleh karena itu, peningkatan yang diamati dalam stabilitas dan aktivitas dapat dipahami dalam hal menstabilkan bentuk aktif enzim, sementara fleksibilitas konformasi yang lebih besar meningkatkan efisiensi katalitik. Namun, studi struktural beresolusi lebih tinggi dan analisis fungsional dengan substrat RNA diperlukan untuk memperjelas efek ini.
Menariknya, peningkatan stabilitas termal dan aktivitas enzimatik E266Q menunjukkan potensi keuntungan dalam replikasi virus. Nsp15 diketahui berperan dalam menghindari respons imun inang dengan mendegradasi intermediet RNA virus bahkan lebih awal yang dapat memicu sensor imun bawaan. Selain itu, ada sedikit pergeseran puncak transisi heksamer di Nsp15-E266Q dibandingkan dengan WT (Gambar 2e ), yang menunjukkan bahwa heksamer Nsp15-E266Q lebih kuat terhadap peningkatan suhu. Ini dapat membantu virus lebih tahan terhadap respons demam inang, yang berpotensi menyebabkan daya infeksi yang lebih tinggi. Sementara E266Q menunjukkan peningkatan sifat enzimatik dan stabilitas, variasi yang diamati dalam tingkat ekspresi membuatnya sulit untuk menghubungkan efek ini hanya dengan mutasi. Penelitian masa depan yang menormalkan ekspresi Nsp15 dalam sistem rekombinan atau konstruksi virus akan diperlukan untuk menentukan apakah perbedaan biokimia ini menghasilkan peningkatan kebugaran virus. Penelitian lebih lanjut diperlukan untuk menetapkan hubungan langsung antara peningkatan sifat biokimia E266Q dan perannya dalam patogenesis virus. Melakukan uji replikasi virus dalam model kultur sel dapat membantu menentukan apakah mutasi ini memberikan keuntungan replikasi. Selain itu, penelitian infeksi in vivo dapat memberikan wawasan tentang mekanisme penghindaran imun. Analisis struktural mutan E266Q dalam kompleks dengan substrat RNA-nya juga dapat mengungkap perubahan mekanistik yang berkontribusi terhadap perubahan aktivitasnya.
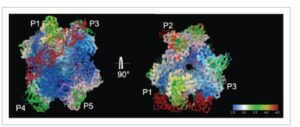
Studi kristalografi sinar-X sebelumnya dari Nsp15 yang dianalisis dalam grup ruang P6 3 , baik dari grup kami maupun grup lain (Jernigan et al. 2023 ; Kim et al. 2020b ), telah secara kuat menunjukkan simetri sterik di seluruh antarmuka dimerik sambil menampilkan asimetri yang menonjol dalam faktor-B. Namun, temuan kami mengungkapkan gerakan asimetris yang melekat pada heksamer Nsp15 WT dan E266Q, sebuah fitur yang sebelumnya tidak jelas dalam studi kristalografi karena simetri yang diprediksi berlebihan. Studi krio-EM terkini menunjukkan bahwa hanya satu monomer per trimer yang mengikat substrat dsRNA (Gambar 5 ) (Frazier et al. 2022a ; Ito et al. 2024 ). Pemrosesan ulang data kami dalam grup ruang simetri rendah mengungkapkan asimetri signifikan dalam fleksibilitas monomer, khususnya terlihat jelas pada mutan E266Q dibandingkan dengan protein WT. Oleh karena itu, asimetri ini mungkin penting untuk mekanisme katalitik protein dan kemungkinan berkontribusi terhadap peningkatan aktivitas enzimatik seperti yang diamati dalam studi biokimia kami.
GAMBAR 5
Buka di penampil gambar
Presentasi PowerPoint
Struktur Cryo-EM dari kompleks Nsp15-dsRNA ( 8UD3 ). Analisis peta kompleks Nsp15-RNA (EMD-42145, konsensus) mengungkapkan pengikatan RNA untai ganda terutama dalam trimer atas (monomer P1 dan P3) dan meluas untuk berinteraksi dengan trimer bawah (monomer P4), seperti yang dilaporkan oleh Ito et al . Analisis resolusi lokal pada tingkat kontur 0,35 mendukung kelengkapan relatif P1 hingga <3,5 Å, termasuk loop, sementara semua loop lain dalam monomer tidak didukung.
Singkatnya, varian alami E266Q dari Nsp15 menggarisbawahi keseimbangan rumit antara aktivitas enzimatik, stabilitas struktural, dan efisiensi ekspresi dalam protein virus. Temuan ini memberikan wawasan berharga tentang mekanisme molekuler yang mendorong evolusi SARS-CoV-2, dengan implikasi untuk mengembangkan strategi terapi yang ditargetkan terhadap garis keturunan Epsilon dan galur baru lainnya.
4 METODE
4.1 Analisis bioinformatika
Sebanyak 126 genom yang spesifik terhadap garis keturunan SARS-CoV-2 Epsilon diunduh dari basis data GISAID (Global Initiative on Sharing Avian Influenza Data) (Shu dan McCauley 2017 ). Genom-genom ini diselaraskan dengan genom referensi Wuhan asli menggunakan Sequencher 5.4.6. Wilayah yang spesifik untuk Nsp15 (koordinat referensi Wuhan 19.621–20.658) dianalisis untuk mengidentifikasi polimorfisme nukleotida tunggal (SNP). SNP yang menyebabkan perubahan asam amino (nsSNP) dalam protein Nsp15 diambil. Skema ditunjukkan pada Gambar S4 .
4.2 Ekspresi Plasmid
Protein Nsp15 yang diekspresikan dirancang dengan tag heksa-histidin yang dapat dibelah TEV (tobacco etch virus) N-terminal. WT diekspresikan dari plasmid pHis-TEV-Nsp15 (Jernigan et al. 2023 ). Plasmid ekspresi yang dihasilkan dalam penelitian ini tercantum dalam Tabel S2 dan dibeli dari GenScript. Urutan DNA plasmid identik dengan pHis-TEV-Nsp15 kecuali untuk perubahan kodon dalam Tabel S2 . Untuk plasmid pHis-TEV-Nsp15-E266Q, Gambar S5 a–c masing-masing menunjukkan peta vektor, urutan DNA lengkap, dan urutan protein yang diekspresikan. Plasmid dan urutannya tersedia dari Addgene (Tabel S2 ).
4.3 Ekspresi dan pemurnian protein
NSSNP WT dan Epsilon Nsp15 diekspresikan dan dimurnikan mengikuti protokol yang telah ditetapkan sebelumnya (Jernigan et al. 2023 ). Gel lengkap disediakan pada Gambar S6 a,b .
4.4 Uji fungsional dan kinetik substrat RNA
Substrat RNA untuk uji fungsional telah dijelaskan sebelumnya (Jernigan et al. 2023 ). Substrat RNA dengan label fluorofor NIR-800 (Near Infra-Red 800nm) pada ujung 3′ disintesis (Integrated DNA Technologies). Oligo RNA dilarutkan pada 100 μM dalam air bebas nuklease dan kemudian diencerkan hingga 5 μM untuk uji fungsional dengan air bebas nuklease.
Pengujian dilakukan dalam 50 mM Tris–HCl pH 7,5, 50 mM KCl, dan 1 mM DTT kecuali dinyatakan lain. Untuk pengujian fungsional, reaksi menggunakan 1,2 μM enzim, 1 μM substrat RNA, dan 5 mM MnCl2 . Campuran reaksi diinkubasi selama 15 menit pada suhu 25°C sebelum diakhiri dengan volume yang sama dari pewarna pemuatan urea 2× (urea 8M, 20 mM Tris–HCl pH 7,5, 1 mM EDTA, 0,05% (b/v) xilena sianol dan 0,05% (b/v) bromofenol biru). Sebelum urea-PAGE, reaksi diinkubasi pada suhu 95°C selama 2 menit. Produk dipisahkan dengan elektroforesis pada urea-PAGE 10% atau 15%. Gel dicitrakan pada ImageQuant 800. Gel lengkap disediakan pada Gambar S6c .
Untuk pengujian kinetik, substrat oligonukleotida RNA fluorogenik 5′6-FAM-dArUdAdA-6-TAMRA3′ dibeli dari GenScript. Pengujian dilakukan dengan 50 nM Nsp15, dan substrat 0,25 μM, yang dilengkapi dengan 5 mM MnCl2 selama 2 jam pada suhu 25 ° C dan dipantau menggunakan filter FAM dalam sistem PCR real-time Stratagene Mx3005P (Agilent Technologies). Untuk Km dan Vmax , pengujian dilakukan dengan konsentrasi substrat berkisar antara 0 hingga 15 μM. Kecepatan awal dihitung dengan perbedaan awal dalam fluoresensi relatif dan waktu. Semua reaksi dilakukan dalam rangkap tiga, dan data dirata-ratakan, diplot, dan dianalisis menggunakan OriginPro 2024b.
4.5 Fluorimetri pemindaian diferensial
Fluorimetri Pemindaian Diferensial dilakukan menurut Kim et al. ( 2020b ). Secara singkat, 10 μM Nsp15 dalam buffer SEC diinkubasi dengan 5X SYPRO Orange selama 30 menit di atas es. Sampel sebanyak 50 μL kemudian dimasukkan ke dalam pelat 96-well pada sistem PCR real-time Vii6 life. Akuisisi sinyal fluoresensi dilakukan dari 20 hingga 95°C dengan laju peningkatan suhu 1°C/60 detik.
4.6 Kristalisasi protein
Kristal Nsp15 diperoleh menggunakan metode batch dengan agitasi, mengikuti protokol yang dijelaskan sebelumnya (Jernigan et al. 2023 ) dengan sedikit modifikasi. Protein Nsp15 yang dimurnikan dalam buffer SEC dipekatkan hingga 15–20 mg/mL (filter sentrifugal Amicon Ultra-0,5 mL dengan MWCO 30 kDa) pada 7000 g dan 12°C. Larutan presipitasi yang mengandung 100 mM HEPES-NaOH, pH 7,5, 200 mM kalsium asetat, dan 8% (b/v) PEG 8000 kemudian dicampur dengan cepat dengan larutan protein pekat di atas es dalam rasio volume 1:6 (protein: presipitasi). Kristal dibiarkan tumbuh pada suhu 4°C.
4.7 Pengumpulan data difraksi sinar-X
4.7.1 Pengaturan Eksperimen
Data difraksi untuk Nsp15-E266Q dikumpulkan pada garis sinar ID29 di Fasilitas Radiasi Sinkrotron Eropa (ESRF). Data diperoleh menggunakan energi sinar-X sebesar 11,56 keV dengan ukuran sinar 4 μm × 2 μm dan paparan 90 μs pada laju pengulangan 231,25 Hz. Transmisi sinar ditetapkan pada 40%, yang memberikan fluks foton rata-rata sebesar 1,44 × 10 11 foton per paparan. Detektor Jungfrau ditempatkan 150 mm dari sampel untuk menangkap sinar-X yang tersebar. Sistem pengiriman target tetap foil kecil khusus yang dikembangkan di ESRF (Doak et al. 2024 ) digunakan, yang membantu mengurangi konsumsi sampel. Sistem ini terdiri dari dua film mylar dengan ketebalan 13 μm. Sekitar 3 μL bubur kristal padat dimasukkan dan disegel di antara dua film mylar, seperti yang dijelaskan dalam Orlans et al. ( 2025 ). Untuk pengumpulan data, foil tersebut dipindai secara terus-menerus dengan pulsa sinar-X pada laju pengulangan 231,25 Hz dengan jarak 20 μm di antara dua bidikan berturut-turut.
4.8 Pengolahan data
4.8.1 Pengolahan dataset Nsp15-E266Q dengan grup spasi P6 3 (Nsp15-E266Q-P6 3 )
Dataset yang diperoleh dari ESRF terdiri dari 48.079 kristal yang ditemukan, yang diidentifikasi menggunakan peakfinder8 dalam rangkaian perangkat lunak CrystFEL (versi 11.1) (White et al. 2012 ). Parameter utama untuk menemukan kristal termasuk ambang batas 800, minimum 10 puncak/gambar, rasio sinyal terhadap derau (SNR) 5,5, batas jumlah piksel berkisar antara 4 hingga 20 piksel/puncak, resolusi minimum (dalam piksel) 60, resolusi maksimum 900, dan jari-jari integrasi 3, 5, dan 7 piksel. Dari 48.079 kristal, 55.068 kristal (114,53%) berhasil diindeks (dengan sel target heksagonal), dengan beberapa pola yang mencerminkan kontribusi dari beberapa kisi kristal. Multi-flag digunakan untuk mendeteksi dan menyelesaikan pola difraksi multi-kisi, memastikan bahwa semua kontribusi dari kristal yang tumpang tindih dalam satu bingkai diindeks secara akurat.
Proses pengindeksan melibatkan kombinasi algoritma, termasuk MOSFLM (Powell 1999 ), XDS (Kabsch 2010 ), Xgandalf (Gevorkov et al. 2019 ), Dirax (Duisenberg 1992 ), ASDF (White 2019 ), dan TakeTwo (Ginn et al. 2016 ). Toleransi pengindeksan ditetapkan pada 10 untuk sumbu sel dan 3,5 untuk sudut antar-aksial. Dataset E266Q-P6 3 menunjukkan ambiguitas pengindeksan. Dengan demikian, intensitas difraksi yang diindeks diproses menggunakan AMBIGATOR (Brehm dan Diederichs 2014 ) (bagian dari paket CrystFEL) dengan operator pengindeksan ulang: -hk,k,-l.
4.8.2 Pengolahan dataset Nsp15-E266Q dengan grup spasi P2 1 (Nsp15-E266Q-P2 1 )
Parameter penemuan dan pengindeksan hit yang sama yang digunakan untuk Nsp15-E266Q dalam grup ruang P6 3 digunakan untuk analisis ini. Pemrosesan data dilakukan menggunakan CrystFEL versi 11.1 (White et al. 2012 ). Sebanyak 48.081 hit diidentifikasi, dan dari jumlah tersebut, 54.091 kristal berhasil diindeks menggunakan sel target monoklinik untuk Nsp15-E266Q.
4.8.3 Pemrosesan ulang dataset WT dengan grup spasi P2 1 (WT-P2 1 )
Percobaan ini dilakukan sebagai bagian dari kampanye RAPID ACCESS X-Ray Free Electron Beamtime selama pandemi COVID-19 (Beamtime P172 dan P182). Jernigan dkk. menjelaskan proses pengiriman sampel dan pengumpulan data secara terperinci (Jernigan dkk. 2023 ). Untuk hit-finding, Peakfinder8 dalam rangkaian perangkat lunak CrystFEL (versi 11.1) digunakan dengan parameter berikut: –threshold = 100 –min-snr = 5 –min-pix-count = 1 –min-res = 80 dan –max-res = 300. Pola difraksi dengan lebih dari 10 puncak diklasifikasikan sebagai hit. Ditemukan 29.674 hit, dan dari hit tersebut, 26.081 kristal diindeks dengan penggunaan gabungan algoritma pengindeksan termasuk MOSFLM (Powell 1999 ), XDS (Kabsch 2010 ), Xgandalf (Gevorkov et al. 2019 ), Dirax (Duisenberg 1992 ), ASDF (White 2019 ), dan TakeTwo (Ginn et al. 2016 ). Sel satuan target adalah sel satuan monoklinik dengan Toleransi pengindeksan ditetapkan pada 10 untuk sumbu sel dan 3,5 untuk sudut antar-aksial.
4.8.4 Penggabungan dan konversi ke format yang kompatibel dengan CCP4
Intensitas refleksi kemudian digabungkan dan diintegrasikan sesuai dengan kelompok titik masing-masing untuk setiap set data dengan PARTIALATOR (juga dari paket CrystFEL), menggunakan satu iterasi dan model kesatuan. Selanjutnya, intensitas yang digabungkan diubah menjadi format mtz yang kompatibel dengan CCP4 (Brehm dan Diederichs 2014 ) menggunakan skrip pembungkus, create-xscale, diikuti oleh XDSCONV (Kabsch 2010 ).
4.9 Penyempurnaan
Kumpulan data berskala diproses menggunakan program AIMLESS (Evans dan Murshudov 2013 ). Untuk penggantian molekuler, program MOLREP (Vagin dan Teplyakov 1997 ) dari paket perangkat lunak CCP4 (Agirre et al. 2023 ) menggunakan struktur kristal Nsp15, khususnya entri PDB 7K9P , sebagai model pencarian. Modifikasi pada model pencarian mencakup penghapusan semua molekul air dan ligan (sitrat). Selain itu, satu rantai dihapus dari model dua rantai awalnya, dan semua faktor B diatur ulang ke 30 Å 2 untuk meminimalkan bias yang diperkenalkan oleh model. Penyempurnaan berikutnya dilakukan menggunakan REFMAC5 (Murshudov et al. 2011 ), Phenix.refine (Afonine et al. 2012 ), dan secara manual melalui COOT (Emsley et al. 2010 ). Terakhir, model tersebut menjalani siklus penyempurnaan akhir dengan jalur PDB-redo (Joosten et al. 2014 ; van Beusekom et al. 2018 ) sebelum diserahkan. Gambar-gambar tersebut diproduksi menggunakan PyMOL (Schrödinger, LLC) (Schrodinger, LLC 2015 ).

